







作者:天羽
在2014年10月河南新乡举办的中国动物学会两栖爬行动物学分会学术研讨会上,东道主河南师范大学对外公布了中国大鲵基因组测序的计划与进展,引起了广泛的关注。为什么大鲵的基因组比较特别呢?其本质的原因就是一个“大”字。据估计,中国大鲵的基因组达到50G左右。如果你对这个数字没有概念,那么没关系,可以和我们人类进行比较,人的基因组一共有2.9G。也就是说,大鲵的基因组大约是人的17倍。更进一步,在人的基因组中,真正编码蛋白质的基因更是少得可怜,大约只占基因组的1%,而这些编码基因在动物当中是比较保守的,也就是说在大鲵中数量应该也差不多。因此,大鲵基因组为什么这么大,成为了长期困扰大家的一个难题。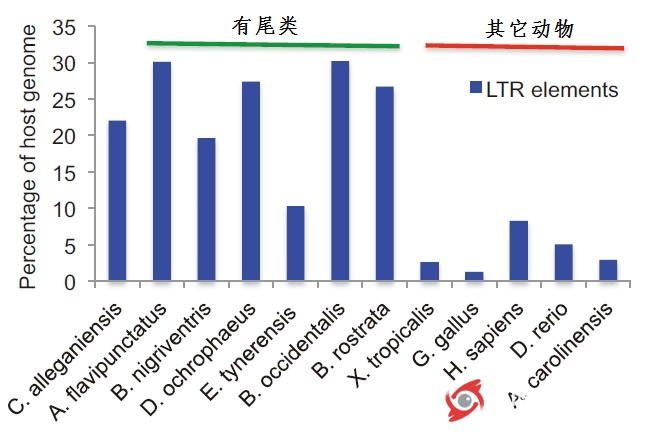
图1. LTR逆转座子在有尾类和其它动物中分布
同时,他们也发现隐鳃鲵基因组小插入缺失的丢失率也比其它动物要低,这也可能是促进有尾类基因组扩张的一个原因。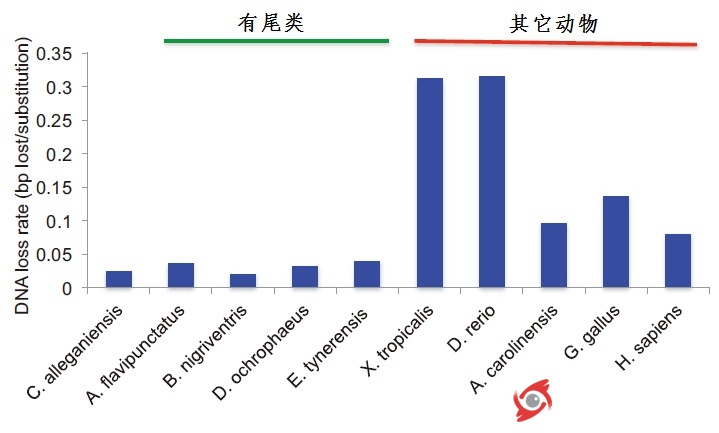
图2. 基因组丢失率的比较
除了基因组的进化,基因组扩张所造成的表型进化可能更值得研究。现在的一些研究认为有尾类动物的一些特征可能与大基因组有关,比如骨骼和循环系统、增大的细胞核和细胞体积、以及降低的细胞分裂分化速率。但是,这些观点大都是基于分析推导而缺少有效证据。对北美隐鳃鲵基因组的研究,向破解动物的大基因组之谜又前进了一步,但要想彻底弄明白这个问题,还需要大量的努力。主管:中国科学院网信办 中国科学院学部工作局 主办:中国科学院计算机网络信息中心
版权所有: 中国科普博览
京ICP备09112257号-28, 43 公网安备11010802031770号
电话:010-58812558 010-58812512